Publication trimestrielle du Laboratoire
d'analyse et d'architecture des systèmes du CNRS

Depuis la découverte de sa structure il y a près de 60 ans, les méthodes pour manipuler et analyser l’ADN n’ont cessé de progresser, ce qui a permis d’aboutir au séquençage complet du génome humain au début des années 2000, et de voir émerger des outils de séquençage haut débit à partir de 2005. Ce dernier exemple de convergence technologique entre la biologie moléculaire et la technologie pour produire des machines d’analyse génomique démontre la vitalité de ce domaine de recherche, et fait nourrir les plus grandes promesses aux yeux des chercheurs et du grand public.
En dépit de ces succès, il n’est pas évident que l’avenir des biotechnologies passe exclusivement par l’industrie du séquençage, en particulier dans son format actuel, comme le démontrent les efforts pour séquencer l’ADN à travers un nanopore ou, plus récemment, par manipulation mécanique. Si l’avenir détient la clé industrielle du séquençage, les travaux de biologie moléculaire des 50 dernières années ont permis de mettre au point une filière industrielle de synthèse d’ADN, des méthodes de modification chimique de l’ADN, et des outils d’amplification et de purification. Ces avancées ouvrent de nouvelles pistes de recherche et développements tout à fait uniques vers une nouvelle ingénierie de technologies ADN dérivées, y compris dans des domaines applicatifs des nanotechnologies non relatifs à la biologie. Une équipe du LAAS s’est pleinement investie de cette problématique et propose quatre axes de contribution.
Ciment programmable pour la nanoconstruction de matériaux avancés
L’idée de détourner la fonction biologique de l’ADN et de l’appréhender comme un matériau pour les nanotechnologies a fait son chemin depuis les années 80, marquées par les travaux exploratoires de Nadrian Seeman sur l’assemblage de structures d’ADN artificielles, et les années 90, où le mariage entre les nanoparticules et l’ADN a été célébré par les équipes de C. Mirkin et P. Alivisatos. L’ADN permet en effet, au travers du jeu de complémentarité des bases le constituant, de programmer et de diriger l’assemblage de structures 1D, 2D ou 3D d’ADN ou bien de nano-objets de nature chimique et de formes variées. Le repliement spécifique de courtes séquences permet de réaliser des édifices de plusieurs centaines de nanomètres (tuiles de Seeman ou Origami ADN), dans lesquels l’organisation est contrôlée depuis l’échelle du nanomètre. L’assemblage de nanoparticules en utilisant l’ADN comme ciment de construction permet aujourd’hui d’assembler des cristaux de nanoparticules aux géométries complexes. Ce travail ouvre la voie à la synthèse de nouveaux matériaux à base de nanoparticules aux propriétés optimisées, comme l’a récemment démontré un travail sur la synthèse de matériaux énergétiques à base de nanoparticules d’aluminium et d’oxyde de cuivre dont la réaction peut être déclenchée à une température de 400°C encore jamais atteinte ou encore la réactivité réglée précisément.
Un matériau malléable pour la reconnaissance moléculaire
L’existence des aptamères révèle une autre facette de l’ADN, mais aussi son potentiel en ingénierie. Grâce aux techniques d’évolution dirigée, fondées sur des cycles de sélection/amplification de l’ADN, il est possible de sélectionner dans une banque d’ADN comprenant ~1015 molécules de séquences distinctes la molécule la plus affine – l’aptamère – pour une cible choisie. En complément du grand nombre de modifications chimiques ou biochimiques accessibles sur les molécules d’ADN, les aptamères offrent des perspectives intéressantes comme couche de détection à adosser à des technologies microsystème pour la fabrication des capteurs du futur. Les travaux actuels de léquipe portent sur deux axes applicatifs. Le premier concerne la détection de la thrombine comme facteur de surveillance de patients atteints de pathologies liées à la coagulation sanguine. Le deuxième est relatif à des problématiques environnementales liées à la détection de polluants chimiques dans les eaux où il s’agit plus précisément de détecter un pesticide organochloré, le métolachlore.
Super-structure reprogrammable pour assurer les fonctions cellulaires
L’ADN génomique est un long fil de 3 milliards de bases replié dans un noyau de quelques dizaines de microns sous la forme de chromosomes. Loin d’être désordonnés, les chromosomes occupent des territoires discrets, et la position des gènes est orchestrée pour faciliter leur expression. Cette super-structure commence tout juste à révéler ses secrets et sa dérégulation apparaît comme un élément clé de nombreuses pathologies, en particulier les cancers. De fait, cette organisation « au-dessus » de l’information génétique, dite épigénétique, fait l’objet d’intenses recherches dont les clés offriront de nouvelles méthodes de diagnostic. Ces recherches sont multi-échelles, puisqu’elles peuvent être menées à l’échelle moléculaire (la paire de base) de la paire de bases ou de la protéine, mais aussi à l’échelle macromoléculaire, en élucidant les lois de repliement dans l’espace des chromosomes, ou encore cellulaire, à travers les motifs d’organisation des chromosomes dans les noyaux. La double contribution du LAAS consiste là à développer de nouveaux outils de manipulation de chromosomes entiers pour étudier les mécanismes de dégradation et de réplication des chromosomes ainsi que des outils d’analyse haut débit du mouvement des chromosomes pour sonder la mécanique des chromosomes dans les cellules vivantes.
“La double contribution du LAAS consiste là à développer de nouveaux outils de manipulation de chromosomes entiers pour étudier les mécanismes de dégradation et de réplication des chromosomes ainsi que des outils d’analyse haut débit du mouvement des chromosomes pour sonder la mécanique des chromosomes dans les cellules vivantes.”
Modéliser l’ADN et ses interactions
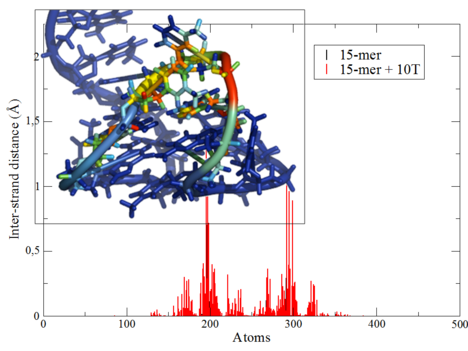
Les technologies ADN représentent un défi méthodologique sans précédent pour la simulation prédictive des procédés d’élaboration et de leurs mécanismes microscopiques associés, au carrefour des technologies bio-hybrides. L’ADN est une structure multi-échelle (de l’ADN au chromosome) qui présente plusieurs niveaux spécifiques de difficulté pour la modélisation : types et nature des interactions intra ou intermoléculaires, flexibilité, nombre d’atomes, espace conformationnel… A cette complexité s’ajoutent les problèmes relatifs à la modélisation des interactions de l’ADN avec des matériaux organiques ou inorganiques non biologiques. Pour répondre à ces exigences de complexité sont mises en œuvre des stratégies de calcul multi-niveaux, c'est-à-dire en combinant plusieurs niveaux de modèles à plusieurs échelles spatiales et temporelles (ab initio, Dynamique Moléculaire…). Une technique originale de calcul est en particulier développée, les Modes Statiques, permettant la prédiction de la flexibilité moléculaire. Ces travaux ont permis d’évaluer et d’optimiser le choix d’espaceurs chimiques nécessaires au greffage de séquences d’ADN sur des substrats organiques ou inorganiques. Ces résultats montrent que nombre de techniques traditionnellement utilisées dans la communauté ne permettent pas de maintenir un repliement optimal de l’ADN ce qui a pour effet d’altérer sa fonction de reconnaissance moléculaire.





